Ansätze
Unter dem Begriff „Synthetische Biologie“ werden Forschungs- und Entwicklungsprojekte mit unterschiedlichen Ansätzen zusammengefasst. Gemeinsam ist ihnen das Ziel, neuartige Organismen oder Komponenten davon zu entwerfen und herzustellen. Sie setzen aber auf unterschiedlichen Ebenen an und weichen auch in ihren Methoden voneinander ab.
Bioengineering
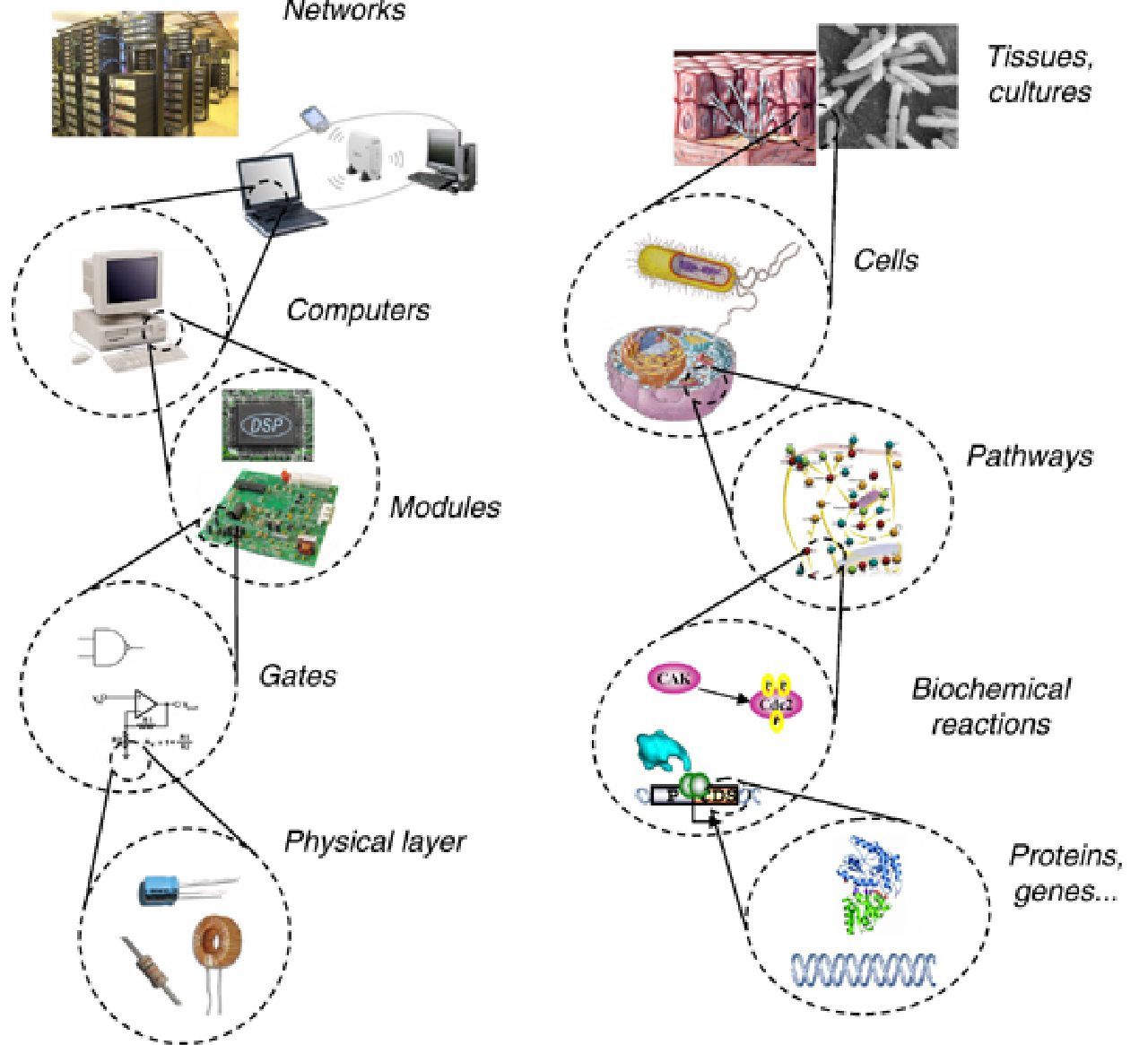
Der Bioengineering-Ansatz ist innerhalb der Synthetischen Biologie vermutlich am weitesten verbreitet. Ihm folgen verschiedene Forschungsgruppen in der Schweiz sowie auch der bekannte „Synthetische Biologie Wettbewerb“ iGEM. Dieser Ansatz wurde anfangs vor allem von Ingenieuren entwickelt. Ihr Ziel ist es, die Biologie zu einer richtigen Ingenieursdisziplin zu machen: Man arbeitet mit standardisierten Bausteinen und kann diese nach einer hierarchischen Struktur zusammenbauen. Standardisierte Bausteine werden nach einem Grundmodell entworfen, das es erlaubt, sie beliebig zu kombinieren. In der Biologie sind diese Bausteine genetische Elemente, die man frei kombinieren kann.
Bevor die Arbeit im Labor beginnt, verbringen Forscher, die sich dieser Form der Synthetischen Biologie verschrieben haben, oft viel Zeit am Computer: sie erstellen detaillierte Modelle von Regulationsmechanismen oder Stoffwechselwegen. Gewisse Forschungsgruppen haben sich sogar ganz auf diesen Aspekt spezialisiert und überlassen anderen Gruppen die Arbeit im Labor. Dieser Ansatz baut auf den Techniken der Gentechnik auf, die Abtrennung zur herkömmlichen Gentechnik ist oft fliessend. Mit der Standardisierung, den Computermodellen und der neuen Dimension von genetischen Veränderungen kommen aber neue Elemente hinzu. Man ist nicht mehr so sehr an die Vorgaben der Natur gebunden, eine neue Dimension von Kreativität wird möglich.
Synthetische Genomik
In der synthetischen Genomik geht es darum, ein gesamtes Erbgut zu synthetisieren, d.h. künstlich im Labor herzustellen. Dabei werden mit Hilfe chemischer und molekularbiologischer Methoden die einzelnen Bausteine der DNA (die Nukleotide) in der gewünschten Reihenfolge aneinandergehängt. Kürzere DNA-Abschnitte – z.B. ein einzelnes Gen – können bereits kommerziell bestellt werden. Aber die künstliche Herstellung von langen Sequenzen wie eines ganzen Erbguts bleibt eine grosse Herausforderung.
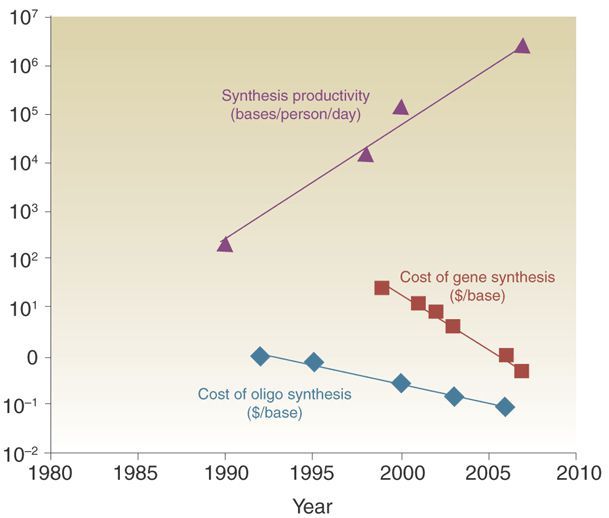
2010 gelang es einer Forschergruppe um Craig Venter, ein Bakterium mit einem komplett synthetisch erzeugten Erbgut herzustellen. Dieses Erbgut wurde nach natürlichem Vorbild produziert, das heisst, es enthielt nahezu die gleiche DNA-Sequenz wie das Erbgut des natürlich vorkommenden Bakteriums. Einige DNA-Abschnitte waren aber vom Menschen entworfen – sie machten es möglich, das Bakterium mit dem künstlichen Genom von natürlichen Exemplaren zu unterscheiden.
Ein Ziel der synthetische Genomik ist es, Genome zu minimieren und unnötige Sequenzen zu entfernen. Diese vereinfachten Genome liessen sich einfacher mit gewünschten Genen und anderen DNA-Sequenzen erweitern.
Xenobiologie
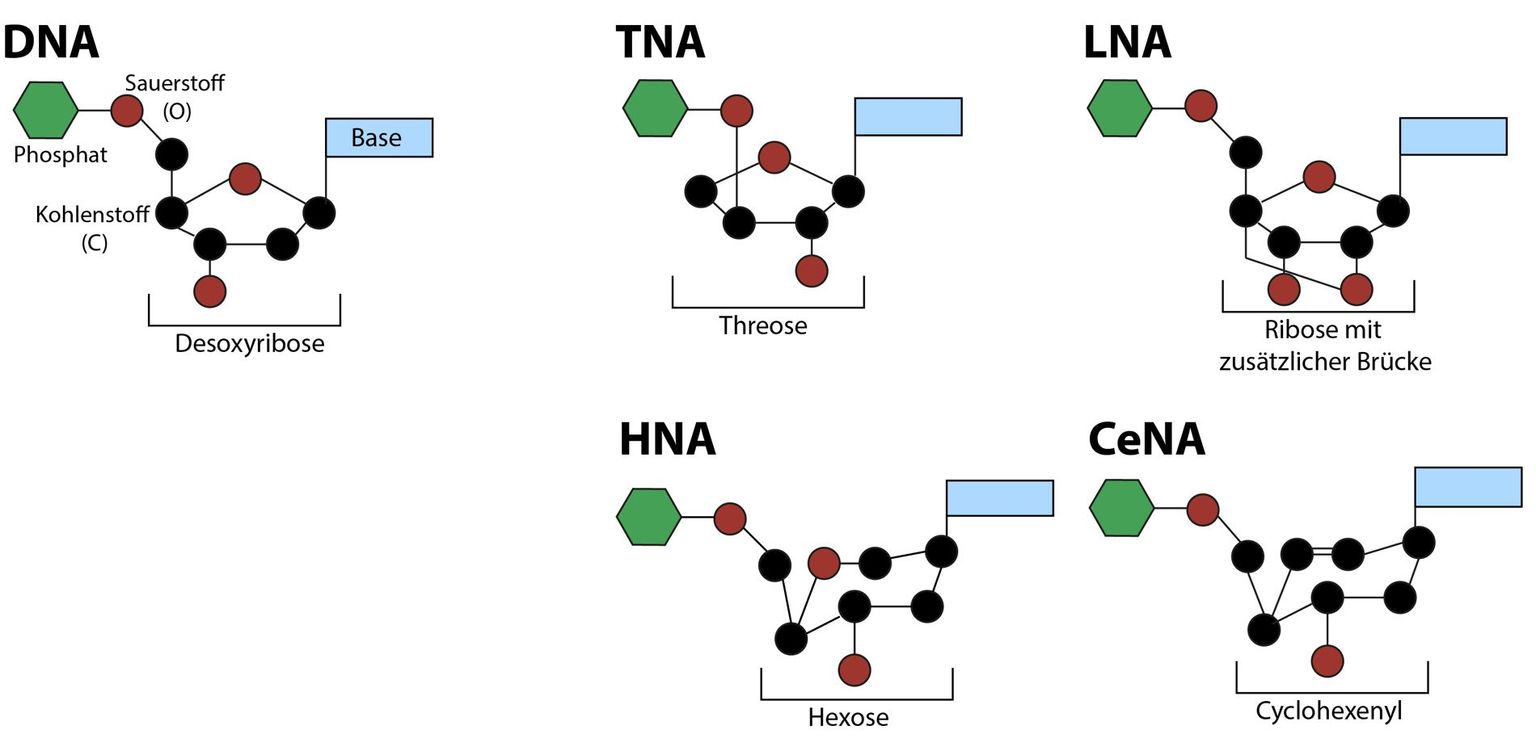
Einen Schritt weiter als in der „synthetische Genomik“ gehen Forscher des Ansatzes, den wir hier Xenobiologie nennen (der aus dem Griechischen stammende Wortteil „xeno“ bedeutet „fremd“ und weist darauf hin, dass diese Biologie so in der Natur nicht vorkommt). Sie haben sich nämlich das Ziel gesetzt, Organismen herzustellen, die ein anderes genetisches System haben als natürliche Lebewesen. Manche Forscher versuchen, neue Formen von Nukleinsäuren als Alternativen zu RNA und DNA zu entwickeln (man spricht von XNA für „xeno nucleic acid“). Andere bleiben bei den herkömmlichen Nukleinsäuren, wollen aber einen neuen genetischen Code entwerfen. Man beginnt damit, in Mikroorganismen, die über ein natürliches Genom verfügen, zusätzlich solche künstlichen Elemente einzufügen. Aber es ist denkbar, dass in ferner Zukunft Organismen entworfen werden könnten, die vollständig auf einem unnatürlichen genetischen System basieren. Der Vorteil solcher Organismen wäre, dass ihre genetischen Elemente nicht durch Viren oder andere Träger auf natürliche Organismen übertragen werden könnten. Denn in natürlichen Organismen würde die molekulare Maschinerie fehlen, um aus diesen künstlichen Genen Eiweisse herzustellen oder im Fall von XNA auch, um sie bei der Zellteilung zu verdoppeln.
Protozellen
Forscher, die diesen Ansatz verfolgen, wollen aus Molekülen lebende Zellen herstellen. Das wären natürlich sehr simple Zellen. Man erhofft sich, daraus zu lernen, wie aus nicht-lebender Materie das Leben auf der Erde entstehen konnte. Als Vorstufen von lebenden Zellen produzieren Forscher sogenannte Protozellen: kleine Bläschen mit einer Fetthülle (wie man sie auch bei natürlichen lebenden Zellen findet), in denen einzelne biochemische Reaktionen ablaufen. Bisher ist man noch weit davon entfernt, Zellen produzieren zu können, die man als „lebend“ bezeichnen würde. Wenn es aber gelingen sollte, solche Zellen herzustellen, wäre es bei dieser Form von Synthetischer Biologie wohl mehr als bei allen anderen angebracht zu sagen, dass man lebende Organismen synthetisiert. Bei allen anderen Ansätzen werden nicht die Mikroorganismen selbst produziert, sondern vorhandenes Erbmaterial wird in andere Mikroorganismen eingefügt.Diese setzen die neue genetische Information um und werden so zu neuartigen Mikroorganismen mit neuen Fähigkeiten und Eigenschaften.
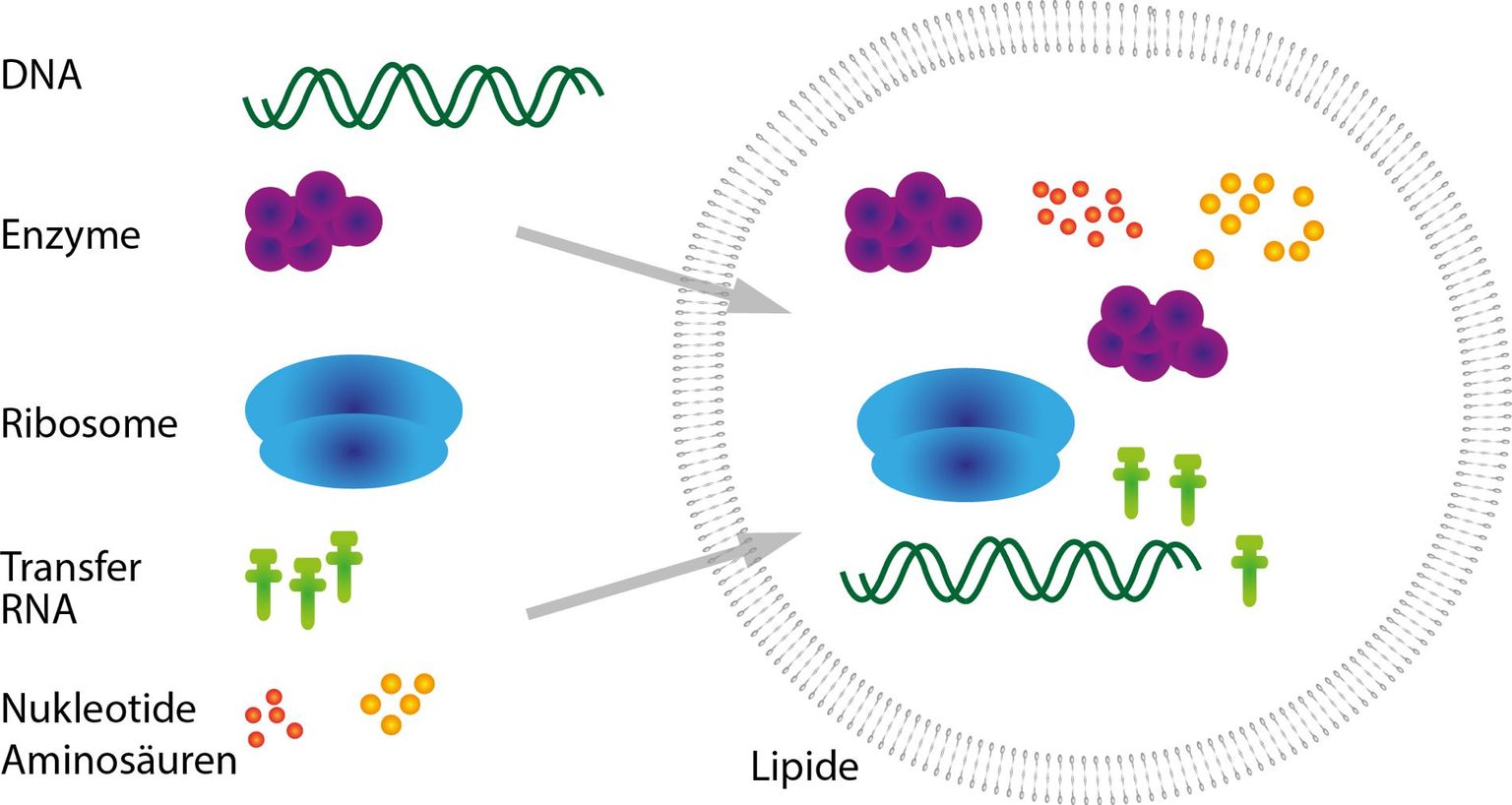
Gemeinsam ist diesen Forschungsprojekten das Ziel, nach einem rationalen, vom Menschen entworfenen «Design» neue Formen von Leben herzustellen. Man kann die verschiedenen Ansätze gröber oder feiner unterteilen, wir haben hier eine Einteilung in vier Ansätze gewählt. Trotz den methodischen und entstehungsgeschichtlichen Unterschieden gibt es Verbindungen zwischen den verschiedenen Ansätzen. So spielt für den Bioengineering-Ansatz der Fortschritt in der DNA-Synthese der „Synthetischen Genomik“ eine wichtige Rolle. Sie ermöglicht es nämlich, neu entworfene DNA-Sequenzen einfach zu produzieren. Es gibt auch Ideen, die künstliche Molekularbiologie mit dem Bioengineering-Ansatz zu verbinden. Für die fernere Zukunft könnte man sich Kombinationen zwischen allen vier Ansätzen vorstellen. Diese könnten es eines Tages gestatten, eine künstliche Protozelle mit einem synthetischen Genom zu entwickeln, die neuartige Stoffwechselwege ermöglicht.
Referenzen
Übersicht
- O’Malley MA et al. (2008) Knowledge-making distinctions in synthetic biology. BioEssays 30: 57-65. Link
- Deplazes A (2009) Piecing together a puzzle. An exposition of synthetic biology. EMBO reports 10: 428-432. Link
Bioengineering
- Endy D (2005) Foundations for engineering biology. Nature 438: 449-453. Link
- Baker D et al. (2006) Engineering Life: Building a FAB for Biology. Scientific American 44-51. Link
Synthetische Genomik
- Gibson DG (2014) Programming biological operating systems: genome design, assembly and activation. Nature Methods 11: 521-526. Link
Xenobiologie
- Benner SA and Sismour AM (2005) Synthetic Biology. Nature Reviews Genetics 6: 533- 543. Link
- Schmidt M (2010) Xenobiology: A new form of life as the ultimate biosafety tool. BioEssays 32: 322-331. Link
Protozellen
- Walde P (2010) Building artificial cells and protocell models: Experimental approaches with lipid vesicles. BioEssays 32: 296-303. Link
- Stano P and Luisi PL (2010) Achievements and open questions in the self-reproduction of vesicles and synthetic minimal cells. Chemical Communications 46: 3639-3653. Link


